Cartographier la diversité de Listeria en abattoir pour comprendre l’origine des souches présentes en transformation
L’Ifip a examiné la diversité de Listeria monocytogenes dans deux abattoirs, pour mieux caractériser les souches présentes sur les différents types de surfaces, leur diversité génétique et leur virulence. Cette fine cartographie permet aux entreprises de mieux surveiller ce germe pathogène et de mettre en place des actions correctives quand cela s’avère nécessaire.
Les travaux de Maury et al. en 2016 ont permis d’identifier des complexes clonaux (CC) avec différents niveaux de virulence. Ainsi, les souches des CC1, CC2, CC4 et CC6 sont hypervirulentes pour l’homme alors que les souches associées aux CC9 et CC121 ont une virulence plus faible. Ce sont justement ces derniers qui sont largement identifiés dans les produits finis de la filière porcine.
L’objectif de ce projet financé par Inaporc était d’identifier la diversité des souches circulant dans le maillon abattage/découpe pour lequel aucune information n’est actuellement disponible en France. Ce projet visait aussi à comprendre l’origine des souches de CC9 et CC121 très prévalentes sur produits finis.
Le Pôle viandes et charcuteries de l’Ifip a réalisé 290 prélèvements dans deux sites d’abattage du Grand Ouest entre mars 2020 et février 2021. Les prélèvements ont été réalisés :
- en zone chaude (abattoir) et en zone froide (découpe/frigo),
- en cours d’activité,
- sur des surfaces en contact ou non avec la matière première.
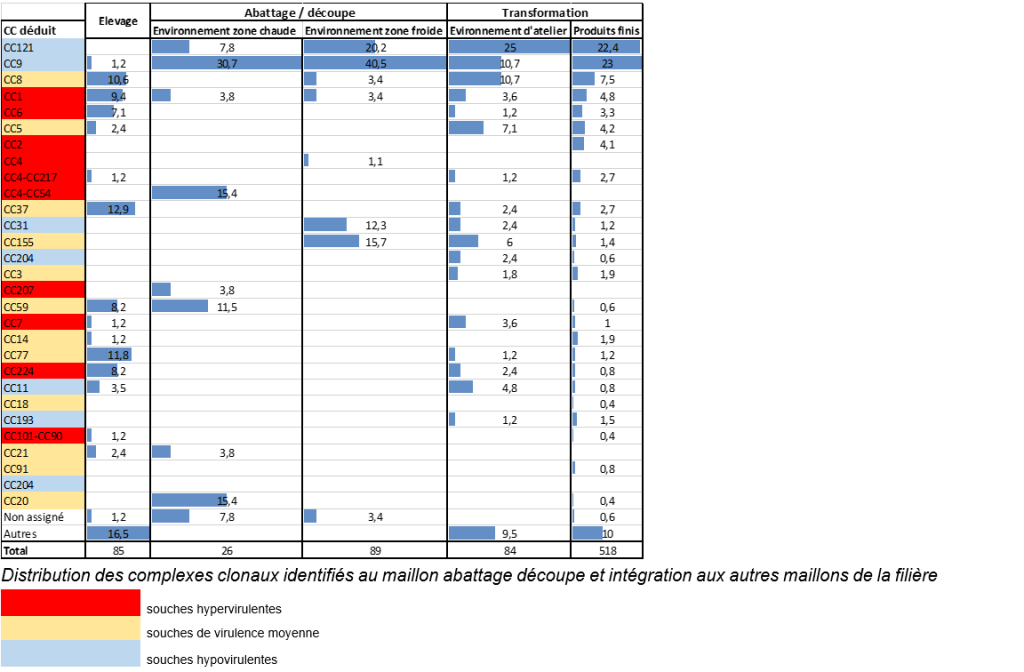
L’Ifip a isolé 115 souches de Listeria monocytogenes, aussi bien en zone chaude qu’en zone froide, correspondant à 12 complexes clonaux différents : 4 hypervirulents, 3 hypovirulents et 5 de virulence moyenne (voir tableau des données de distribution intégrant les résultats précédents dans les autres maillons de la filière publiés par Félix et al., 2018).
En zone chaude, la diversité des complexes clonaux était un peu plus forte qu’en zone froide et les souches hypervirulentes plus fréquentes (20,8% contre 4,6% en zone froide). Aucune souche de complexe clonal hypervirulent n’a été identifiée sur les surfaces sans contact avec la matière première (zones de persistance potentielle de souches).
Les souches de CC9 (38%) et CC121 (17%) étaient les plus identifiées dans notre étude. Absentes ou quasi absentes en élevage, elles apparaissaient en zone chaude à l’abattage et s’implantaient fortement en zone froide (découpe/frigo) puis en transformation de produits. Les souches de CC9 apparaissaient après abattage, lors de la fente de la carcasse. Les souches de CC121 étaient présentes, même si faiblement, dans les siphons de l’abattoir principalement.
Pour chaque site d’abattage, la diversité des types génétiques des souches de Listeria monocytogenes isolées a été établie et a permis d’identifier des souches persistantes et de mettre en place des mesures correctives. Ces données de diversité des complexes clonaux seront consolidées grâce aux prélèvements supplémentaires actuellement effectués sur d’autres sites d’abattage. L’identification de souches hypervirulentes des complexes clonaux CC1, CC2, CC4, CC6 sur les outils de découpe, notamment sur les surfaces sans contact avec la matière première, doit entraîner des mesures de nettoyage/désinfection renforcées dans les entreprises.
Etude financée par Inaporc.